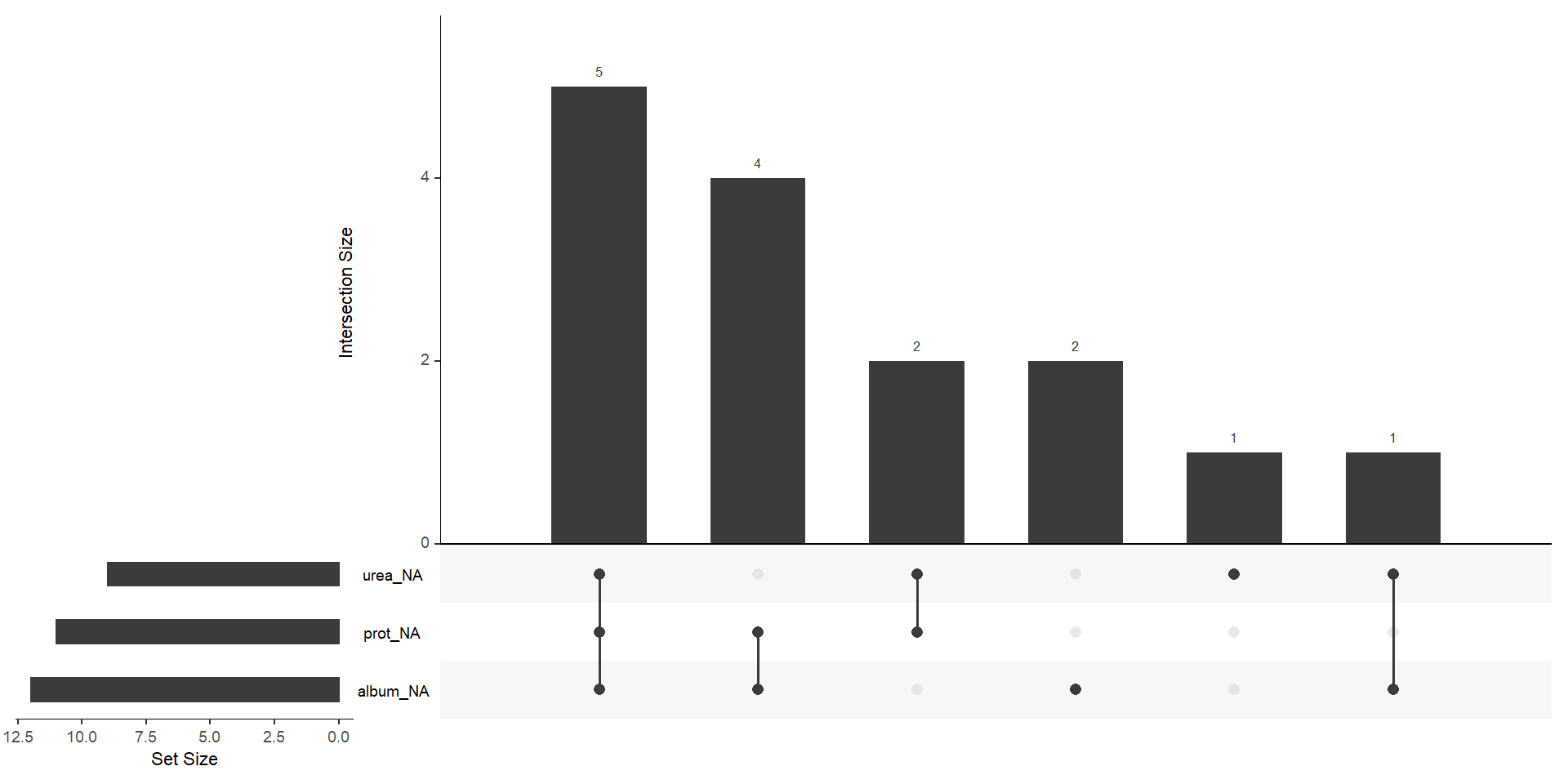Sesión 4
Curso: R Aplicado a los Proyectos de Investigación
Percy Soto-Becerra, M.D., M.Sc(c)
InkaStats Data Science Solutions | Medical Branch
2022-10-10

Manejo de datos 2: Categorización y etiquetado de variables
Agenda
Manejo de datos 2: Categorización y etiquetado de variables
AID / AED en R
Creación de variables con case_when()
Función de apoyo a mutate()para crear variables según condiciones más complejas.
Crea variables de acuerdo a condiciones complejas
- Toda transformación o creación de variables en dplyr ocurre por mutate; por tanto, case_when()se utiliza dentro de un mutate
datos %>%
mutate(
nueva_var = case_when(
condición1 ~ resultado1,
condición2 ~ resultado2,
condición3 ~ resultado3
)
)- Atajo de teclado para obtener
~: Alt + 1 + 2 + 6
Condición no cumplida en case_when()
- Para indicar que todas las condiciones previas no se cumplen, se debe colocar
TRUE:
datos %>%
mutate(
nueva_var = case_when(
condición1 ~ resultado1,
condición2 ~ resultado2,
condición3 ~ resultado3,
TRUE ~ resultado_si_condición_no_se_cumple
)
)Es muy importante siempre colocarla al final de cualquier conjunto de condiciones previas.
Cuando el resultado de no cumplirse es que se asigne valor perdido, es importante usar la función
as.tipo_var()sobre el indicador de dato perdidoNA.Si es character: as.character(NA)
Si es numeric: as.numeric(NA)
case_when() en acción
- Se quieren crear categorías de edad: “20-30”, “31-35” y “36-41”
datos_fase1 %>%
select(id, age) %>%
mutate(agecat = case_when(age >= 20 & age <= 30 ~ "20-30",
age >= 31 & age <= 35 ~ "31-35",
age >= 36 & age <= 41 ~ "36-41",
TRUE ~ as.character(NA))
) # A tibble: 106 × 3
id age agecat
<dbl> <dbl> <chr>
1 1 33 31-35
2 1 32 31-35
3 2 27 20-30
4 2 27 20-30
5 3 25 20-30
6 3 25 20-30
7 4 37 36-41
8 4 38 36-41
9 5 31 31-35
10 5 32 31-35
# … with 96 more rows- Se desea crear variable indicadora de inclusión en estudio: Solo pacientes sin pareja y que proceden de Callao, Carabayllo, Chorrillos o SJL son elegibles:
datos_fase1 %>%
mutate(elegible = case_when(
married2 == "Without couple" & procedence %in% c("Callao", "Carabayllo", "Chorrillos", "SJL") ~ "Elegible",
TRUE ~ "No elegible"
)
) # A tibble: 106 × 15
id time treat age race married marri…¹ proce…² weight height e2
<dbl> <fct> <fct> <dbl> <chr> <fct> <fct> <chr> <dbl> <dbl> <dbl>
1 1 Baseline Place… 33 Mest… Single Withou… Callao 59 1.4 87.3
2 1 3 months Place… 32 Mest… Single Withou… Callao 59.9 1.3 210.
3 2 Baseline Dosis… 27 Mest… Single Withou… Santa … 62 1.5 169.
4 2 3 months Dosis… 27 Mest… Single Withou… Santa … 62.1 1.6 99.9
5 3 Baseline Dosis… 25 Mest… Single Withou… Callao 62 1.6 78.8
6 3 3 months Dosis… 25 Mest… Single Withou… Callao 60 1.6 155.
7 4 Baseline Dosis… 37 Mest… Divorc… Withou… Callao 60.9 1.5 41.0
8 4 3 months Dosis… 38 Mest… Divorc… Withou… Callao 61.4 1.5 109.
9 5 Baseline Place… 31 Mest… Single Withou… La Mol… 64 1.5 43.0
10 5 3 months Place… 32 Mest… Single Withou… La Mol… 58.1 1.6 56.0
# … with 96 more rows, 4 more variables: lh <dbl>, fsh <dbl>, prog <dbl>,
# elegible <chr>, and abbreviated variable names ¹married2, ²procedenceEtiquetar variables con set_var_labels()
La función set_var_labels() del paquete labelled() es muy útil para etiquetar columnas.
Los datos deben tener metadatos que permitan ser legibles por el ser humano.
Primero instalar y cargar paquete:
- Es preferible usar esta función al final de todo el proceso de limpieza de datos.
set_var_labels() en acción
Nuestro turno
Descargue la carpeta denominada taller04 disponible en la carpeta compartida.
Abra el proyecto denominado taller04.Rproj
Complete y ejecute el código faltante en los chunk de código de la PRIMERA PARTE.
Una vez culmine todo el proceso, renderice el archivo .qmd.
Hagamos una pausa
Tomemos un descanso de 5 minutos…
Estire las piernas …
Deje de ver las pantallas …
… cualquier , las del celular también.
05:00
AID / AED en R
Agenda
Manejo de datos 2: Categorización y etiquetado de variables
AID / AED en R
Paso 1: Resumen global de los datos
Dimensiones: columnas y filas
Variables y tipos
Datos completos y faltantes
Variables numéricas: Mínimos, máximos y valores extremos
Variables categóricas: Valores o categorías muy poco frecuentes y datos perdidos encubiertos
- Heche un vistazo de los datos con glimpse():
- La función skim() del paquete {skimr} genera un resumen global de los datos:
| Name | datos |
| Number of rows | 3 |
| Number of columns | 3 |
| _______________________ | |
| Column type frequency: | |
| numeric | 3 |
| ________________________ | |
| Group variables | None |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
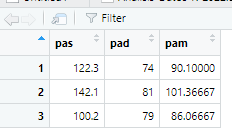
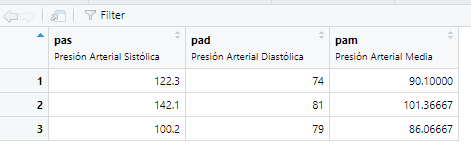
| pas | 0 | 1 | 121.53 | 20.96 | 100.20 | 111.25 | 122.3 | 132.20 | 142.10 | ▇▁▇▁▇ |
| pad | 0 | 1 | 78.00 | 3.61 | 74.00 | 76.50 | 79.0 | 80.00 | 81.00 | ▇▁▁▇▇ |
| pam | 0 | 1 | 92.51 | 7.93 | 86.06 | 88.08 | 90.1 | 95.74 | 101.37 | ▇▇▁▁▇ |
- La función describe() del paquete {Hmisc} genera un reporte general bien detallado, variable por variable:
datos
3 Variables 3 Observations
--------------------------------------------------------------------------------
pas
n missing distinct Info Mean Gmd
3 0 3 1 121.5 27.93
Value 100.2 122.3 142.1
Frequency 1 1 1
Proportion 0.333 0.333 0.333
--------------------------------------------------------------------------------
pad
n missing distinct Info Mean Gmd
3 0 3 1 78 4.667
Value 74 79 81
Frequency 1 1 1
Proportion 0.333 0.333 0.333
--------------------------------------------------------------------------------
pam
n missing distinct Info Mean Gmd
3 0 3 1 92.51 10.21
Value 86.06 90.10 101.37
Frequency 1 1 1
Proportion 0.333 0.333 0.333
--------------------------------------------------------------------------------Paso 2: Detecte y maneje duplicados
La función get_dupes() del paquete {janitor} es útil para esto.
Si solo colocamos get_dupes(), entonces nos identifica duplicados de fila completa:
Si colocamos una o más variables dentro de get_dupes(), entonces nos identifica duplicados solo de esa variable.
A menudo lo hacemos para encontrar individus duplicados.
Si el duplicado es erróneo, lo podemos eliminar con distinct() y el argumento .keep_all = TRUE.
Se debde espeficiar si el duplicado es de fila o de alguna variable (p. ej., id).
id_jaula id_raton tratamiento protocolo peso_inicial
1 1 1 control ovx 26.00
2 1 2 control ovx 24.50
3 1 3 control ovx 20.40
4 2 4 control hemiovx 26.59
5 2 5 control ovx 23.50
6 2 6 maca ovx 25.00
7 2 7 maca ovx 24.80
8 3 8 maca ovx 23.20
9 3 9 maca hemiovx 22.69
10 3 10 maca ovx 23.90
11 5 11 maca + critro ovx 21.90
12 5 12 maca + critro ovx 23.40
13 5 13 maca + critro ovx 21.90
14 5 14 maca + critro ovx 22.40
15 8 15 triple dosis maca + citro ovx 18.90
16 8 16 triple dosis maca + citro ovx 23.50
17 9 17 triple dosis maca + citro ovx 24.50
18 9 18 triple dosis maca + citro ovx 25.30
19 9 19 triple dosis maca + citro ovx 27.90
20 9 20 sham operated no ovx 25.50
21 10 21 sham operated no ovx 23.40
22 10 22 sham operated no ovx 22.50
23 10 23 sham operated no ovx 22.90
peso_final peso_utero chol glucose tag prot urea album
1 33.28 0.089 85.990 109.97 182.42 5.37 66.27 66.82
2 30.50 0.063 94.460 81.62 211.87 4.68 76.73 55.36
3 29.93 0.078 99.670 118.37 195.16 NA 52.32 NA
4 32.19 0.134 83.380 71.91 98.46 NA 50.71 NA
5 30.37 0.052 82.080 95.53 108.13 5.33 26.02 NA
6 30.43 0.055 107.490 160.36 141.10 NA NA 72.14
7 28.77 0.064 76.870 195.53 95.82 5.02 40.78 67.09
8 27.30 0.062 95.760 182.41 105.49 6.12 66.94 70.64
9 26.86 0.070 85.340 184.51 90.99 NA NA 66.27
10 29.10 0.113 82.080 168.50 143.30 4.85 34.48 83.73
11 24.93 0.108 80.781 113.38 128.35 NA NA NA
12 26.70 0.022 88.590 102.62 282.64 NA NA NA
13 28.67 0.014 104.880 60.10 200.00 5.33 37.96 NA
14 28.37 0.046 70.350 150.13 199.12 4.93 45.61 64.64
15 23.80 0.057 78.170 110.50 114.73 4.78 38.23 52.77
16 26.70 0.065 85.340 144.88 134.51 5.15 43.06 59.59
17 28.77 0.016 64.400 138.85 160.44 NA NA NA
18 29.20 0.068 67.100 140.68 127.03 NA NA NA
19 29.48 0.076 65.790 114.70 169.67 6.17 NA NA
20 30.27 0.088 74.260 94.23 185.05 4.93 NA 68.59
21 27.90 0.535 59.280 126.77 244.84 NA 60.37 NA
22 26.68 0.081 68.400 78.22 93.19 NA 157.89 NA
23 27.43 0.176 84.690 125.20 108.13 NA NA NA¿Qué pasa si no se sabe si el duplicado es erróneo?
Podemos tener dos o más filas con duplicados y no saber cuál es el correcto.
En estos casos, el problema es complejo. Una solución puede ser la deduplicación probabilística.
Paso 3: Identifique datos faltantes
Evalúe número y porcentaje de datos perdidos así como el patrón de estos.
Hay varios paquetes que permiten manejar datos perdidos:
- {VIM}
- {visdat}
- {naniar}
- {otros}
Usaremos algunas funciones de {visdat}, {VIM}y {naniar}.
{visdat}y {nanair}generan gráficos {ggplot2}, mientras que {VIM} no lo hace.
Paso 3: Identifique datos faltantes (cont.)
- Nuevamente skim() nos permite conocer, rápidamente, el número de datos perdidos.
| Name | datos |
| Number of rows | 23 |
| Number of columns | 13 |
| _______________________ | |
| Column type frequency: | |
| character | 2 |
| numeric | 11 |
| ________________________ | |
| Group variables | None |
Variable type: character
| skim_variable | n_missing | complete_rate | min | max | empty | n_unique | whitespace |
|---|---|---|---|---|---|---|---|
| tratamiento | 0 | 1 | 4 | 25 | 0 | 5 | 0 |
| protocolo | 0 | 1 | 3 | 7 | 0 | 3 | 0 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| id_jaula | 0 | 1.00 | 5.30 | 3.38 | 1.00 | 2.00 | 5.00 | 9.00 | 10.00 | ▇▃▅▂▇ |
| id_raton | 0 | 1.00 | 12.00 | 6.78 | 1.00 | 6.50 | 12.00 | 17.50 | 23.00 | ▇▆▇▆▇ |
| peso_inicial | 0 | 1.00 | 23.68 | 1.99 | 18.90 | 22.59 | 23.50 | 24.90 | 27.90 | ▂▃▇▇▂ |
| peso_final | 0 | 1.00 | 28.59 | 2.18 | 23.80 | 27.08 | 28.77 | 30.10 | 33.28 | ▂▆▇▅▂ |
| peso_utero | 0 | 1.00 | 0.09 | 0.10 | 0.01 | 0.06 | 0.07 | 0.09 | 0.54 | ▇▁▁▁▁ |
| chol | 0 | 1.00 | 81.96 | 12.89 | 59.28 | 72.31 | 82.08 | 87.29 | 107.49 | ▅▃▇▃▃ |
| glucose | 0 | 1.00 | 124.74 | 37.27 | 60.10 | 99.08 | 118.37 | 147.50 | 195.53 | ▅▇▇▃▅ |
| tag | 0 | 1.00 | 153.06 | 52.36 | 90.99 | 108.13 | 141.10 | 190.11 | 282.64 | ▇▃▅▁▂ |
| prot | 11 | 0.52 | 5.22 | 0.49 | 4.68 | 4.91 | 5.08 | 5.34 | 6.17 | ▇▃▅▁▃ |
| urea | 9 | 0.61 | 56.95 | 32.34 | 26.02 | 38.87 | 48.16 | 64.80 | 157.89 | ▇▃▁▁▁ |
| album | 12 | 0.48 | 66.15 | 8.44 | 52.77 | 62.12 | 66.82 | 69.62 | 83.73 | ▃▃▇▂▂ |
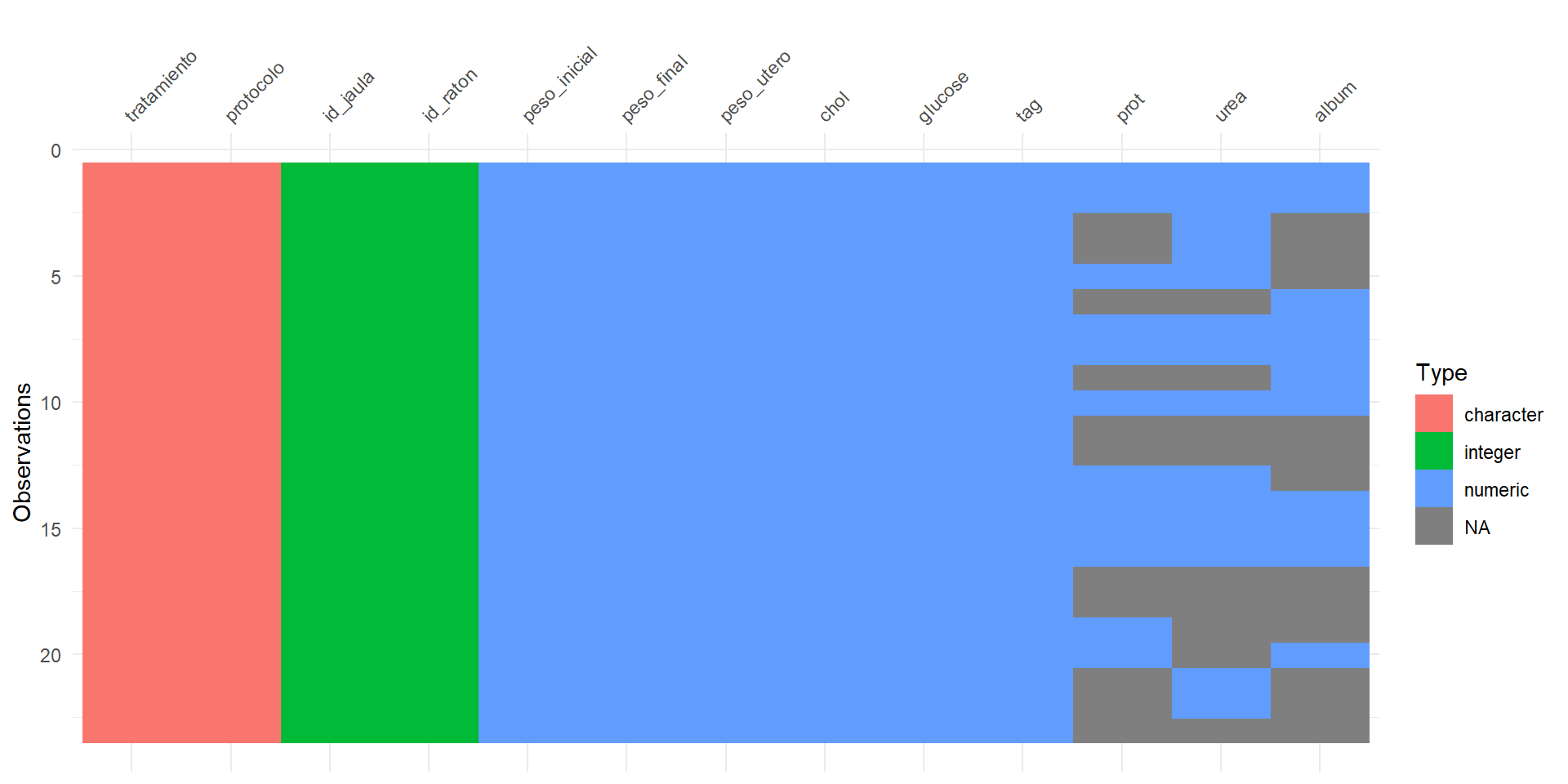
- El paquete {visdat} te permite visualizar el tipo de dato y si hay o no presencia de datos perdidos
Es importante verificar si el tipo de dato corresponde con la naturaleza de la variable de estudio.
Algunos datos faltantes pueden no verse por no configurar apropiadamente el tipo de la variable.
- Podemos también generar gráficos para identificar los datos perdidos y sus combinaciones:
Se aprecia que la variable
prottiene 47.83% de sus datos faltantes. La variableureatiene 39.13% de sus datos faltantes.La legenda que dice
Missing (10.7%)indica que el total de datos faltantes en las celdas (no en las filas) es de 10.7%.¿Cuántos datos faltantes en por fila tendremos? ¿Qué combinaciones de datos faltantes tendremos?
Paso 3: Identifique datos faltantes (cont.)
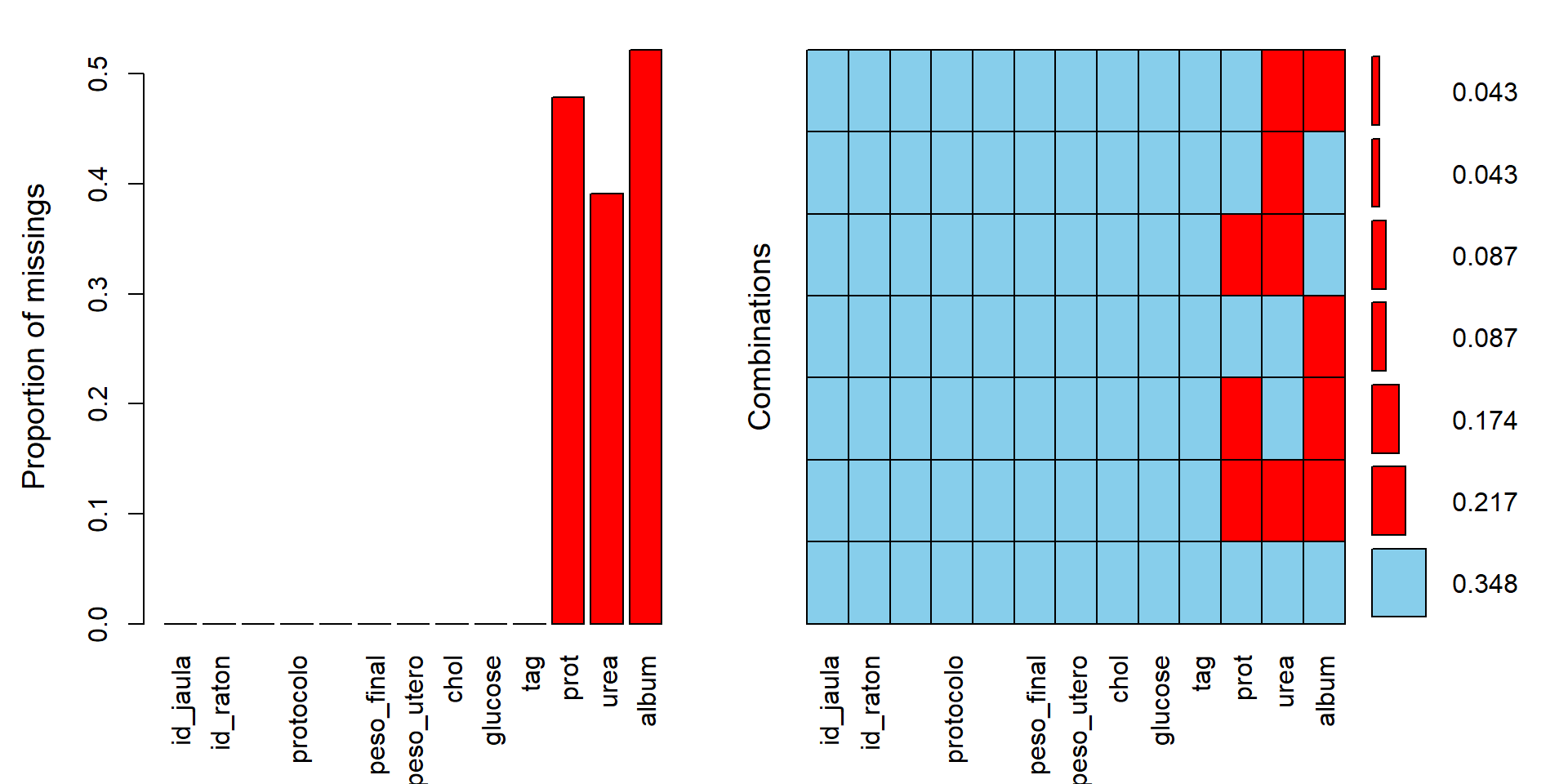
El paquete {VIM} permite identificar datos perdidos por variable y sus combinaciones.
Podemos visualizar los resultados directamente con la función aggr():
- También podemos usar la función gg_miss_upset del paquete {naniar} para evaluar las combinaciones de datos perdidos:
Lo primero que uno debe tratar de hacer es recuperar los datos faltantes.
Volver a revisar documentos fuentes.
Recontactar sujetos, etc.
Podemos identificar a los individuos con datos faltantes en la variable urea usando filter():
id_jaula id_raton urea
1 2 6 NA
2 3 9 NA
3 5 11 NA
4 5 12 NA
5 9 17 NA
6 9 18 NA
7 9 19 NA
8 9 20 NA
9 10 23 NASi se recupera la información, uno puede remplazar los valores usando código en R.
La función replace()del paquete {dplyr} es útil para esto. Supongamos que el dato perdido para el ratón 6 es de 65.2, podemos rempalzar el dato usando replace()
id_jaula id_raton urea
1 1 1 66.27
2 1 2 76.73
3 1 3 52.32
4 2 4 50.71
5 2 5 26.02
6 2 6 65.20
7 2 7 40.78
8 3 8 66.94
9 3 9 NA
10 3 10 34.48
11 5 11 NA
12 5 12 NA
13 5 13 37.96
14 5 14 45.61
15 8 15 38.23
16 8 16 43.06
17 9 17 NA
18 9 18 NA
19 9 19 NA
20 9 20 NA
21 10 21 60.37
22 10 22 157.89
23 10 23 NALos datos perdidos a veces se guardan por defecto con algunos caracteres especiales.
Pueden ser problemáticas si se guardan con categorías como: -99, 8888, “No aplica”, “No sabe”, etc.
Una función muy útil para lidiar con estos datos y convertirlos en
NAes la función replace_na()del paquete {tidyr}
edad diabetes
1 45 Sí
2 23 Sí
3 34 No
4 29 N/A
5 -999 No
6 23 Sí
7 34 No
8 57 N/A
9 88 N/A
10 -999 N/A
11 -999 Sí- Podemos convertir directamente todos estos valores por default a datos perdidos:
Paso 4: Identifique valores extremos no plausibles
- Revise, variable por variable
valores extremos no plausiblesoplausibles, pero sospechosamente extremos. El valor mínimo esp0y el valor máximo esp100. Deben ser plausibles.
| Name | datos |
| Number of rows | 23 |
| Number of columns | 13 |
| _______________________ | |
| Column type frequency: | |
| character | 2 |
| numeric | 11 |
| ________________________ | |
| Group variables | None |
Variable type: character
| skim_variable | n_missing | complete_rate | min | max | empty | n_unique | whitespace |
|---|---|---|---|---|---|---|---|
| tratamiento | 0 | 1 | 4 | 25 | 0 | 5 | 0 |
| protocolo | 0 | 1 | 3 | 7 | 0 | 3 | 0 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| id_jaula | 0 | 1.00 | 5.30 | 3.38 | 1.00 | 2.00 | 5.00 | 9.00 | 10.00 | ▇▃▅▂▇ |
| id_raton | 0 | 1.00 | 12.00 | 6.78 | 1.00 | 6.50 | 12.00 | 17.50 | 23.00 | ▇▆▇▆▇ |
| peso_inicial | 0 | 1.00 | 23.68 | 1.99 | 18.90 | 22.59 | 23.50 | 24.90 | 27.90 | ▂▃▇▇▂ |
| peso_final | 0 | 1.00 | 28.59 | 2.18 | 23.80 | 27.08 | 28.77 | 30.10 | 33.28 | ▂▆▇▅▂ |
| peso_utero | 0 | 1.00 | 0.09 | 0.10 | 0.01 | 0.06 | 0.07 | 0.09 | 0.54 | ▇▁▁▁▁ |
| chol | 0 | 1.00 | 81.96 | 12.89 | 59.28 | 72.31 | 82.08 | 87.29 | 107.49 | ▅▃▇▃▃ |
| glucose | 0 | 1.00 | 124.74 | 37.27 | 60.10 | 99.08 | 118.37 | 147.50 | 195.53 | ▅▇▇▃▅ |
| tag | 0 | 1.00 | 153.06 | 52.36 | 90.99 | 108.13 | 141.10 | 190.11 | 282.64 | ▇▃▅▁▂ |
| prot | 11 | 0.52 | 5.22 | 0.49 | 4.68 | 4.91 | 5.08 | 5.34 | 6.17 | ▇▃▅▁▃ |
| urea | 9 | 0.61 | 56.95 | 32.34 | 26.02 | 38.87 | 48.16 | 64.80 | 157.89 | ▇▃▁▁▁ |
| album | 12 | 0.48 | 66.15 | 8.44 | 52.77 | 62.12 | 66.82 | 69.62 | 83.73 | ▃▃▇▂▂ |
- Permite hacer algo similar
datos
13 Variables 23 Observations
--------------------------------------------------------------------------------
id_jaula
n missing distinct Info Mean Gmd
23 0 7 0.979 5.304 3.881
lowest : 1 2 3 5 8, highest: 3 5 8 9 10
Value 1 2 3 5 8 9 10
Frequency 3 4 3 4 2 4 3
Proportion 0.130 0.174 0.130 0.174 0.087 0.174 0.130
--------------------------------------------------------------------------------
id_raton
n missing distinct Info Mean Gmd .05 .10
23 0 23 1 12 8 2.1 3.2
.25 .50 .75 .90 .95
6.5 12.0 17.5 20.8 21.9
lowest : 1 2 3 4 5, highest: 19 20 21 22 23
--------------------------------------------------------------------------------
tratamiento
n missing distinct
23 0 5
lowest : control maca maca + critro sham operated triple dosis maca + citro
highest: control maca maca + critro sham operated triple dosis maca + citro
control (5, 0.217), maca (5, 0.217), maca + critro (4, 0.174), sham operated
(4, 0.174), triple dosis maca + citro (5, 0.217)
--------------------------------------------------------------------------------
protocolo
n missing distinct
23 0 3
Value hemiovx no ovx ovx
Frequency 2 4 17
Proportion 0.087 0.174 0.739
--------------------------------------------------------------------------------
peso_inicial
n missing distinct Info Mean Gmd .05 .10
23 0 19 0.998 23.68 2.244 20.55 21.90
.25 .50 .75 .90 .95
22.59 23.50 24.90 25.90 26.53
lowest : 18.90 20.40 21.90 22.40 22.50, highest: 25.30 25.50 26.00 26.59 27.90
Value 18.90 20.40 21.90 22.40 22.50 22.69 22.90 23.20 23.40 23.50 23.90
Frequency 1 1 2 1 1 1 1 1 2 2 1
Proportion 0.043 0.043 0.087 0.043 0.043 0.043 0.043 0.043 0.087 0.087 0.043
Value 24.50 24.80 25.00 25.30 25.50 26.00 26.59 27.90
Frequency 2 1 1 1 1 1 1 1
Proportion 0.087 0.043 0.043 0.043 0.043 0.043 0.043 0.043
--------------------------------------------------------------------------------
peso_final
n missing distinct Info Mean Gmd .05 .10
23 0 21 0.999 28.59 2.483 25.11 26.68
.25 .50 .75 .90 .95
27.08 28.77 30.10 30.49 32.02
lowest : 23.80 24.93 26.68 26.70 26.86, highest: 30.37 30.43 30.50 32.19 33.28
--------------------------------------------------------------------------------
peso_utero
n missing distinct Info Mean Gmd .05 .10
23 0 23 1 0.0927 0.07728 0.0166 0.0268
.25 .50 .75 .90 .95
0.0560 0.0680 0.0885 0.1298 0.1718
lowest : 0.014 0.016 0.022 0.046 0.052, highest: 0.108 0.113 0.134 0.176 0.535
--------------------------------------------------------------------------------
chol
n missing distinct Info Mean Gmd .05 .10
23 0 21 0.999 81.96 14.9 64.54 66.05
.25 .50 .75 .90 .95
72.31 82.08 87.29 98.89 104.36
lowest : 59.28 64.40 65.79 67.10 68.40, highest: 94.46 95.76 99.67 104.88 107.49
--------------------------------------------------------------------------------
glucose
n missing distinct Info Mean Gmd .05 .10
23 0 23 1 124.7 43.44 72.54 78.90
.25 .50 .75 .90 .95
99.08 118.37 147.50 179.63 184.30
lowest : 60.10 71.91 78.22 81.62 94.23, highest: 160.36 168.50 182.41 184.51 195.53
--------------------------------------------------------------------------------
tag
n missing distinct Info Mean Gmd .05 .10
23 0 22 1 153.1 59.41 93.45 96.35
.25 .50 .75 .90 .95
108.13 141.10 190.11 209.50 241.54
lowest : 90.99 93.19 95.82 98.46 105.49, highest: 199.12 200.00 211.87 244.84 282.64
--------------------------------------------------------------------------------
prot
n missing distinct Info Mean Gmd .05 .10
12 11 10 0.993 5.222 0.5367 4.735 4.787
.25 .50 .75 .90 .95
4.910 5.085 5.340 6.045 6.143
lowest : 4.68 4.78 4.85 4.93 5.02, highest: 5.15 5.33 5.37 6.12 6.17
Value 4.68 4.78 4.85 4.93 5.02 5.15 5.33 5.37 6.12 6.17
Frequency 1 1 1 2 1 1 2 1 1 1
Proportion 0.083 0.083 0.083 0.167 0.083 0.083 0.167 0.083 0.083 0.083
--------------------------------------------------------------------------------
urea
n missing distinct Info Mean Gmd .05 .10
14 9 14 1 56.95 30.41 31.52 35.52
.25 .50 .75 .90 .95
38.87 48.16 64.80 73.79 105.14
lowest : 26.02 34.48 37.96 38.23 40.78, highest: 60.37 66.27 66.94 76.73 157.89
Value 26.02 34.48 37.96 38.23 40.78 43.06 45.61 50.71 52.32
Frequency 1 1 1 1 1 1 1 1 1
Proportion 0.071 0.071 0.071 0.071 0.071 0.071 0.071 0.071 0.071
Value 60.37 66.27 66.94 76.73 157.89
Frequency 1 1 1 1 1
Proportion 0.071 0.071 0.071 0.071 0.071
--------------------------------------------------------------------------------
album
n missing distinct Info Mean Gmd .05 .10
11 12 11 1 66.15 9.592 54.06 55.36
.25 .50 .75 .90 .95
62.12 66.82 69.62 72.14 77.94
lowest : 52.77 55.36 59.59 64.64 66.27, highest: 67.09 68.59 70.64 72.14 83.73
Value 52.77 55.36 59.59 64.64 66.27 66.82 67.09 68.59 70.64 72.14 83.73
Frequency 1 1 1 1 1 1 1 1 1 1 1
Proportion 0.091 0.091 0.091 0.091 0.091 0.091 0.091 0.091 0.091 0.091 0.091
--------------------------------------------------------------------------------El
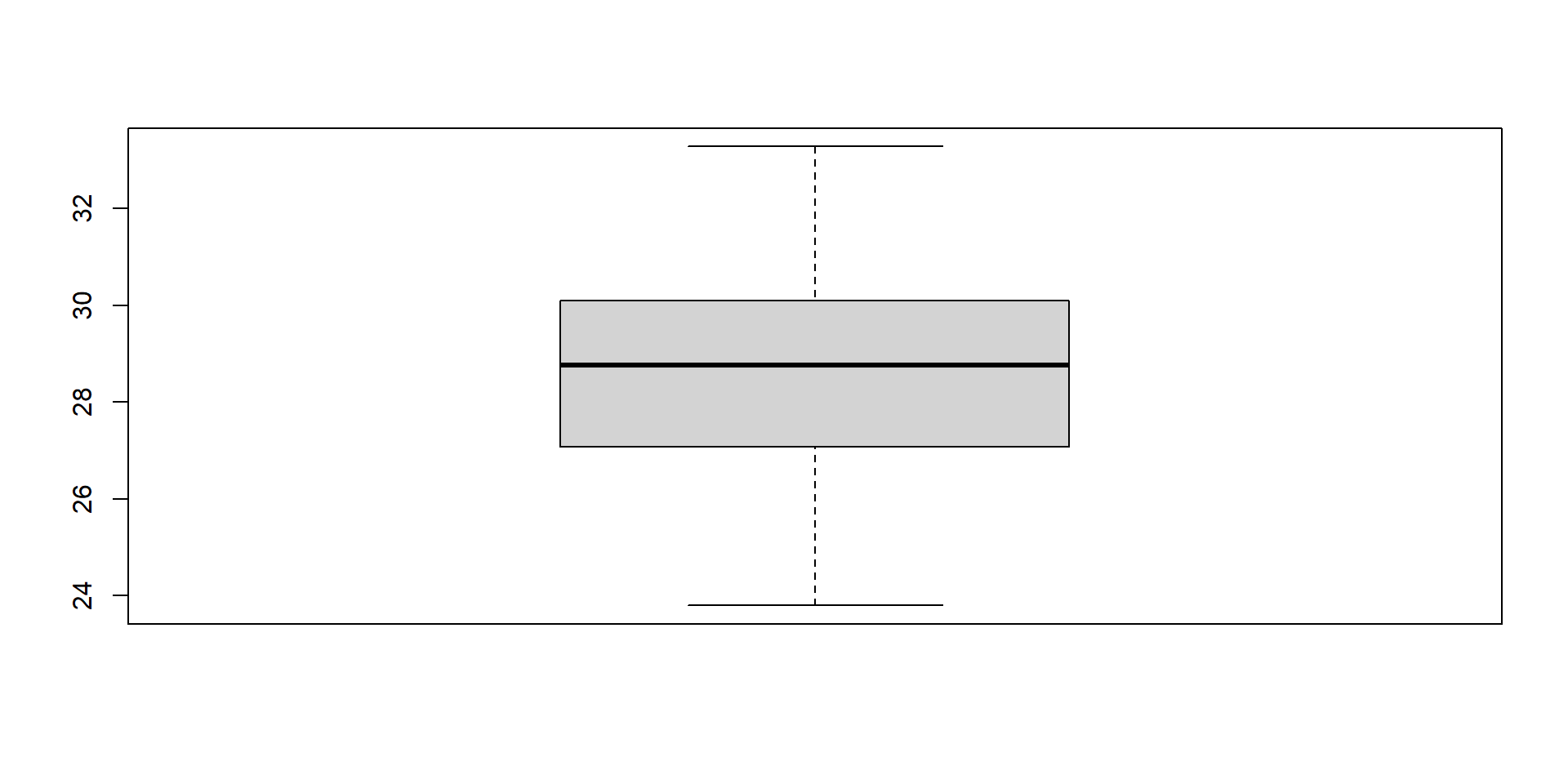
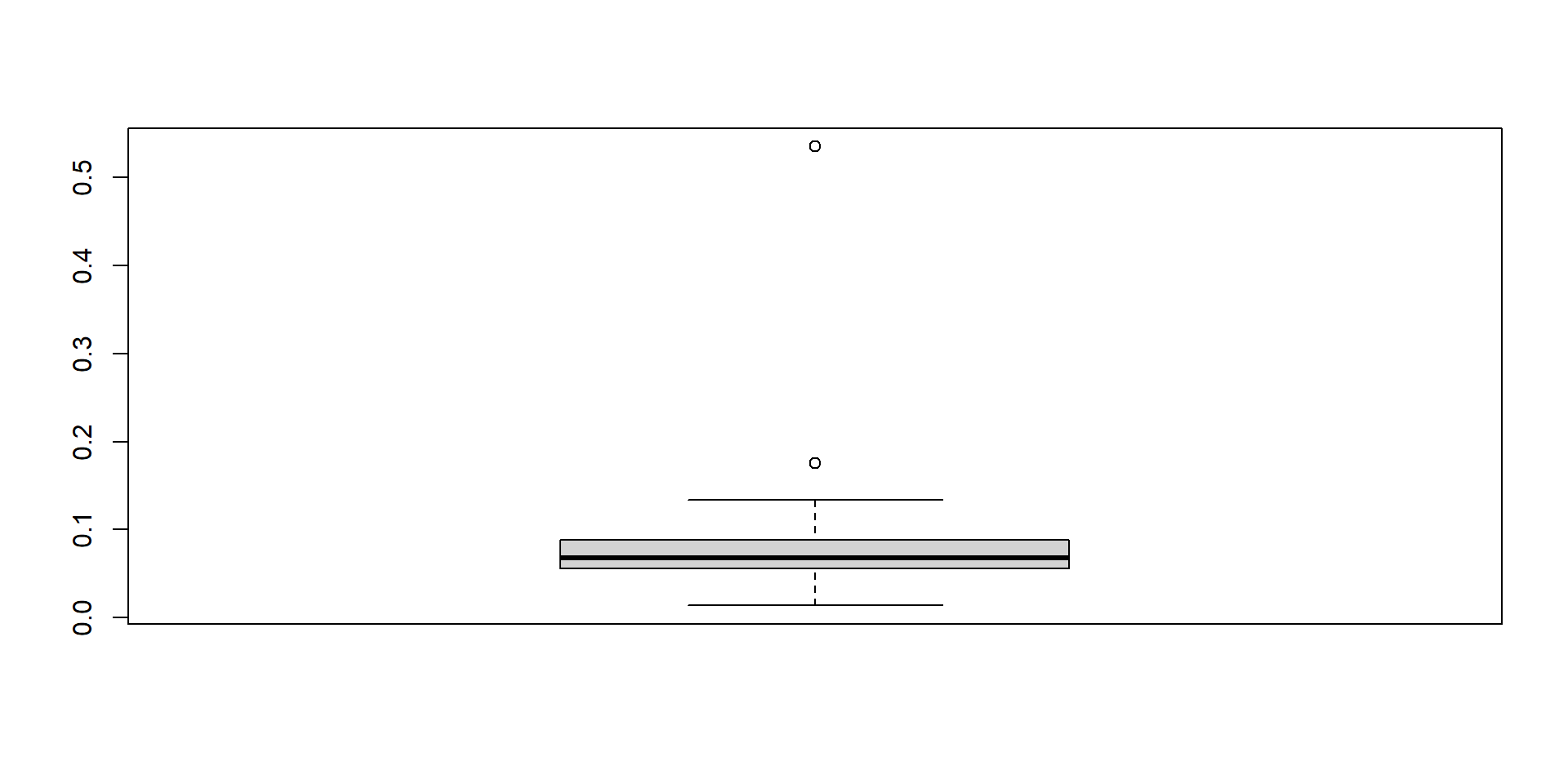
gráfico de cajasnos muestra la disrtibución de la variable numérica en termino de sus cuantiles.Los
puntos aislados, fuera de las cajas y bigotes, son considerados valores extremos.Estos pueden ser
plausiblesono plausibles.El gráfico de cajas permite
identificar, rápidamente,valores extremospotencialmenteno plausiblesoproblemáticos.
Los datos extremos pueden ser valores anómalos válidos.
En ocasiones, son valores no plausibles, inválidos, producto del mal recojo de información.
Cuando se tenga valores extremos no plausibles se puede optar por dos acciones:
- Corregir el valor extremo no plausible por datos que sí sean plausibles.
- Si no se puede, convertir los valores extremos no plausibles en datos faltantes (veremos esto).
- Bonus: A veces puede ser mejor recortar los datos y quedarse con el 1% y 99% percentil más bajo y alto, respectivamente.
Paso 4: Identifique valores extremos no plausibles
- Se puede usar la función: na_if() del paquete {dplyr}.
Veamos una base de datos juguete con datos de peso (kg) y hemoglobina (mg/dL) de pacientes en un estudio:
El peso de 1450 es un valor extremo no plausible. Igualmente, los valores de hemoglobina 213, 3124 y -4 son valores extremos no plausibles.
Lo primero que debemos hacer es recuperar es tratar de recuperar estos valores.
Supongamos que podemos recuperar los valores: 1450 en realidad es 45 kg; 213, 3124 y -4 son 11.3, 10.44 y 9.2 mg/dL.
Podemos usar la función recode para corregir los valores de peso:
peso hb
1 56 12
2 34 11
3 23 213
4 78 10
5 46 3124
6 45 -4- También podemos corregir de varias variables simultáneamente:
- Si no podemos recuperar los datos correctos, la otra opción es convertir los valores extremos en datos faltantes:
peso hb
1 56 12
2 34 11
3 23 213
4 78 10
5 46 3124
6 NA -4- Podemos hacerlo de manera simultánea para varias variables
datos_extremo %>%
mutate(
peso = na_if(peso, 1450),
hb = na_if(hb, 213),
hb = na_if(hb, 3124),
hb = na_if(hb, -4)
) peso hb
1 56 12
2 34 11
3 23 NA
4 78 10
5 46 NA
6 NA NA- O usando replace() y una condición lógica:
Paso 5: Detecte y corrija inconsistencias mediante consultas (queries) de interés
Muestre el peso inicial mínimo, máximo y promedio del grupo control:
datos %>% filter(tratamiento == "control") %>% summarise( minimo_peso = min(peso_inicial), maximo_peso = max(peso_inicial), promedio_peso = mean(peso_inicial) )datos %>% filter(tratamiento == "control") %>% summarise( minimo_peso = min(peso_inicial), maximo_peso = max(peso_inicial), promedio_peso = mean(peso_inicial) )datos %>% filter(tratamiento == "control") %>% summarise( minimo_peso = min(peso_inicial), maximo_peso = max(peso_inicial), promedio_peso = mean(peso_inicial) )
minimo_peso maximo_peso promedio_peso
1 20.4 26.59 24.198Muestre los pesos inicial máximos, mínimo y promedio según grupo de tratamiento. También muestre el número de ratones por grupo:
datos %>% group_by(tratamiento) %>% summarise( minimo_peso = min(peso_inicial), maximo_peso = max(peso_inicial), promedio_peso = mean(peso_inicial), n_ratones = n() )datos %>% group_by(tratamiento) %>% summarise( minimo_peso = min(peso_inicial), maximo_peso = max(peso_inicial), promedio_peso = mean(peso_inicial), n_ratones = n() )datos %>% group_by(tratamiento) %>% summarise( minimo_peso = min(peso_inicial), maximo_peso = max(peso_inicial), promedio_peso = mean(peso_inicial), n_ratones = n() )
# A tibble: 5 × 5
tratamiento minimo_peso maximo_peso promedio_peso n_ratones
<chr> <dbl> <dbl> <dbl> <int>
1 control 20.4 26.6 24.2 5
2 maca 22.7 25 23.9 5
3 maca + critro 21.9 23.4 22.4 4
4 sham operated 22.5 25.5 23.6 4
5 triple dosis maca + citro 18.9 27.9 24.0 5Muestre los id_jaula con el número de ratones por jaula
datos %>% group_by(id_jaula) %>% summarise(n_ratones_por_jaula = n())datos %>% group_by(id_jaula) %>% summarise(n_ratones_por_jaula = n())datos %>% group_by(id_jaula) %>% summarise(n_ratones_por_jaula = n())
# A tibble: 7 × 2
id_jaula n_ratones_por_jaula
<int> <int>
1 1 3
2 2 4
3 3 3
4 5 4
5 8 2
6 9 4
7 10 3Identifique los ID de los ratones del grupo control con una razón glucosa / colesterol > 1
datos %>% filter(tratamiento == "control" & glucose / chol > 1)datos %>% filter(tratamiento == "control" & glucose / chol > 1)
id_jaula id_raton tratamiento protocolo peso_inicial peso_final peso_utero
1 1 1 control ovx 26.0 33.28 0.089
2 1 3 control ovx 20.4 29.93 0.078
3 2 5 control ovx 23.5 30.37 0.052
chol glucose tag prot urea album
1 85.99 109.97 182.42 5.37 66.27 66.82
2 99.67 118.37 195.16 NA 52.32 NA
3 82.08 95.53 108.13 5.33 26.02 NAOtra forma de hacerlo, es crear primerio la razón glucose / chol y filtrar:
datos %>% mutate(ratio_gluc_chol = glucose / chol) %>% filter(tratamiento == "control" & ratio_gluc_chol > 1)datos %>% mutate(ratio_gluc_chol = glucose / chol) %>% filter(tratamiento == "control" & ratio_gluc_chol > 1)datos %>% mutate(ratio_gluc_chol = glucose / chol) %>% filter(tratamiento == "control" & ratio_gluc_chol > 1)
id_jaula id_raton tratamiento protocolo peso_inicial peso_final peso_utero
1 1 1 control ovx 26.0 33.28 0.089
2 1 3 control ovx 20.4 29.93 0.078
3 2 5 control ovx 23.5 30.37 0.052
chol glucose tag prot urea album ratio_gluc_chol
1 85.99 109.97 182.42 5.37 66.27 66.82 1.278870
2 99.67 118.37 195.16 NA 52.32 NA 1.187619
3 82.08 95.53 108.13 5.33 26.02 NA 1.163865Nuestro turno
Descargue la carpeta denominada taller04 disponible en la carpeta compartida.
Abra el proyecto denominado taller04.Rproj
Complete y ejecute el código faltante en los chunk de código de la SEGUNDA PARTE.
Una vez culmine todo el proceso, renderice el archivo .qmd.
10:00
@psotob91
https://github.com/psotob91
percys1991@gmail.com
R Aplicado a los Proyectos de Investigación - Sesión 4