Sesión 9
Curso: R Aplicado a los Proyectos de Investigación
Percy Soto-Becerra, M.D., M.Sc(c)
InkaStats Data Science Solutions | Medical Branch
2022-10-21

El Modelo Lineal Generalizado
Agenda
El Modelo Lineal Generalizado
La regresión de Poisson
La regresión Binomial Negativa
Tablas de regresión reproducibles con {gtsummary}
Modelo Lineal Generalizado
Modelo lineal que permite modelar desenlaces de varios tipos de distribuciones.
Generaliza el modelo de regresión lineal.
Permite que Yi siga otras distribuciones.
- Pertenecientes a la familia de distribución exponencial.
Modelo Lineal Generalizado: Anatomía
Componente sistemático:
g(E(Y|x1i,...,xpi))=g(E(Yi))=ηi=β0+β1x1i+...+βpxip
g() es la
función de enlace.ηi es el
predictor linear.E(Y|x1i,...,xpi)=μi
Componente aleatorio:
Yi∼Distribucion de la Familia Exponencial
Familia exponencial
| Variable respuesta | Distribución de FE | Función de enlace canónica g() | Otras funciones de enlace comunes |
|---|---|---|---|
| Binaria | Bernoulli (Binomial con n = 1) | logit() | log() |
| Conteo | Binomial (con n > 1) | logit() | log() |
| Poisson | log() | ||
| Binomial negativo | log(μ+k) | log() | |
| Continua positiva | Gamma | 1μ | |
| Gausiana inversa |
Estimación de GLM
Hace uso de
estimación de máxima verosimilitud(MV).Salvo el caso normal (donde MV = MCO),
no existe solución cerradapara obtener los estimadores de MV.- Hay que hacer uso de
métodos numéricos: Fisher Scoring, Newton Raphson, etc.
- Hay que hacer uso de
No siempre la función de verosimilitud tiene un máximo.
Solo cuando se usa la función de enlace canónica.
Caso contrario, puede no tener solución única y hay problemas de convergencia.
La regresión de Poisson
Agenda
El Modelo Lineal Generalizado
La regresión de Poisson
La regresión Binomial Negativa
Tablas de regresión reproducibles con {gtsummary}
El modelo de regresión de Poisson
yi∼Poisson(β0+β1x1i+...+βpxip)
- Componente sistemático:
log(E(yi))=ηi
- Función de enlace:
ηi=β0+β1x1i+...+βpxip
- Componente aleatorio:
yi∼Poisson(ηi)
¿Por qué usar log?
log() es la función de enlace canónica: solución única para MV y no problemas de convergencia por esto.
Si usamos la función identidad de la regresión lineal, el modelo quedaría planetado de esta manera:
E(yi)=β0+β1x1i+...+βpxip
- Entonces, el modelo predecirá valores fuera del rango natural de la variable yi:
- yi es de conteo (discreto), pero se obtendrían predichos con decimales (continuo).
- yi es positivo siempre, pero se podrían ontener predichos negativos.
- El inverso de log(), exp() devuelve una
razón de medias, medida interpretable.
¿Por qué no asumir normalidad de yi?
Porque la distribución de yi no es normal, es una variable de conteo.
El principal problema de esto, es que al ser Poisson, la media=varianza=λ, por lo que a mayor valor de la media, la varianza aumentará.
Lo que implica que yi es una v.a. heterocedástica.
El modelo normal necesita homocedasticidad, caso contrario, tiene que corregirse de alguna manera.
Poisson no necesita esto, su modelo es heterocedastico por naturaleza, lo que hace más eficiente la estimación:
- Si el modelo es válido, los intervalos de confianza serán más precisos.
La regresión de Poisson retorna razón de medias
La regresión de Poisson permite retornar directamente
razón de medias(RM).Los coeficientes de regresión β del modelo son log(RM), por lo tanto, podemos exponenciarlos para obtener los OR:
β=log(RM)
eβ=RM
glm() paso a paso
Call:
glm(formula = docvis ~ female + age, data = md_visit)
Deviance Residuals:
Min 1Q Median 3Q Max
-5.139 -2.792 -1.623 0.593 118.711
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -0.442309 0.168002 -2.633 0.00848 **
female 0.980325 0.082751 11.847 < 2e-16 ***
age 0.071883 0.003682 19.522 < 2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for gaussian family taken to be 33.29453)
Null deviance: 671518 on 19608 degrees of freedom
Residual deviance: 652772 on 19606 degrees of freedom
AIC: 124390
Number of Fisher Scoring iterations: 2Se especifica la ecuación.
- Por defecto, regresión lineal normal
glm() paso a paso
Call:
glm(formula = docvis ~ female + age, family = poisson, data = md_visit)
Deviance Residuals:
Min 1Q Median 3Q Max
-3.3562 -2.2285 -1.1346 0.3396 26.8860
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -0.0368083 0.0176597 -2.084 0.0371 *
female 0.3092910 0.0081168 38.105 <2e-16 ***
age 0.0227500 0.0003624 62.768 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for poisson family taken to be 1)
Null deviance: 122270 on 19608 degrees of freedom
Residual deviance: 116363 on 19606 degrees of freedom
AIC: 153636
Number of Fisher Scoring iterations: 6Se indica la familia de distribución de yi.
- Por defecto, la función de enlace será la canónica.
glm() paso a paso
Call:
glm(formula = docvis ~ female + age, family = poisson(link = "log"),
data = md_visit)
Deviance Residuals:
Min 1Q Median 3Q Max
-3.3562 -2.2285 -1.1346 0.3396 26.8860
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -0.0368083 0.0176597 -2.084 0.0371 *
female 0.3092910 0.0081168 38.105 <2e-16 ***
age 0.0227500 0.0003624 62.768 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for poisson family taken to be 1)
Null deviance: 122270 on 19608 degrees of freedom
Residual deviance: 116363 on 19606 degrees of freedom
AIC: 153636
Number of Fisher Scoring iterations: 6Se indica la familia de distribución de yi.
- Por defecto, la función de enlace será la canónica.
glm() paso a paso
mod <- glm(docvis ~ female + age,
family = poisson(link = "identity"),
data = md_visit)
summary(mod)
Call:
glm(formula = docvis ~ female + age, family = poisson(link = "identity"),
data = md_visit)
Deviance Residuals:
Min 1Q Median 3Q Max
-3.1714 -2.2486 -1.1320 0.3312 26.8394
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -0.18852 0.04629 -4.073 4.64e-05 ***
female 1.00553 0.02510 40.056 < 2e-16 ***
age 0.06581 0.00110 59.848 < 2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for poisson family taken to be 1)
Null deviance: 122270 on 19608 degrees of freedom
Residual deviance: 116471 on 19606 degrees of freedom
AIC: 153743
Number of Fisher Scoring iterations: 6- Podemos cambiar la función de enlace.
glm() paso a paso
mod <- glm(docvis ~ female + age,
family = poisson(link = "log"),
data = md_visit)
mod %>%
tidy() %>%
gt()| term | estimate | std.error | statistic | p.value |
|---|---|---|---|---|
| (Intercept) | -0.03680829 | 0.0176596601 | -2.084315 | 0.03713153 |
| female | 0.30929097 | 0.0081167612 | 38.105220 | 0.00000000 |
| age | 0.02275001 | 0.0003624464 | 62.767938 | 0.00000000 |
- En el caso de la función de enlace log, los coeficientes son
log(razón de medias), para volverlos interpretables, hay que aplicar antilogaritmo:exp().
glm() paso a paso
| term | estimate | std.error | statistic | p.value |
|---|---|---|---|---|
| (Intercept) | 0.9638609 | 0.0176596601 | -2.084315 | 0.03713153 |
| female | 1.3624587 | 0.0081167612 | 38.105220 | 0.00000000 |
| age | 1.0230108 | 0.0003624464 | 62.767938 | 0.00000000 |
- La función tidy() de {broom} permite hacer esto facilmente:
exponentiate = TRUE.
glm() paso a paso
| term | estimate | std.error | statistic | p.value | conf.low | conf.high |
|---|---|---|---|---|---|---|
| (Intercept) | 0.9638609 | 0.0176596601 | -2.084315 | 0.03713153 | 0.9310334 | 0.9977672 |
| female | 1.3624587 | 0.0081167612 | 38.105220 | 0.00000000 | 1.3409630 | 1.3843148 |
| age | 1.0230108 | 0.0003624464 | 62.767938 | 0.00000000 | 1.0222845 | 1.0237380 |
- También podemos agregar intervalos de confianza.
Casos aplicado
- Identificar factores asociados a que el niño tenga alergia.
- Factores asociados al número de visitas médicas anuales.
- Especificación del modelo
Call:
glm(formula = docvis ~ female + age, family = poisson(link = "log"),
data = md_visit)
Deviance Residuals:
Min 1Q Median 3Q Max
-3.3562 -2.2285 -1.1346 0.3396 26.8860
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -0.0368083 0.0176597 -2.084 0.0371 *
female 0.3092910 0.0081168 38.105 <2e-16 ***
age 0.0227500 0.0003624 62.768 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for poisson family taken to be 1)
Null deviance: 122270 on 19608 degrees of freedom
Residual deviance: 116363 on 19606 degrees of freedom
AIC: 153636
Number of Fisher Scoring iterations: 6- Presentación con intervalos de confianza y exponenciada (OR):
female: El número medio de visitas anuales al médico en mujeres fue 20% veces más el de los hombres (RM = 1.33; IC95% 1.31 a 1.35; p < 0.001)age: Por cada incremento de la edad en un año, el número medio de visitas anuales al médico se incrementa en 1% (RM = 1.017; IC95% 1.016 a 1.018; p < 0.001).
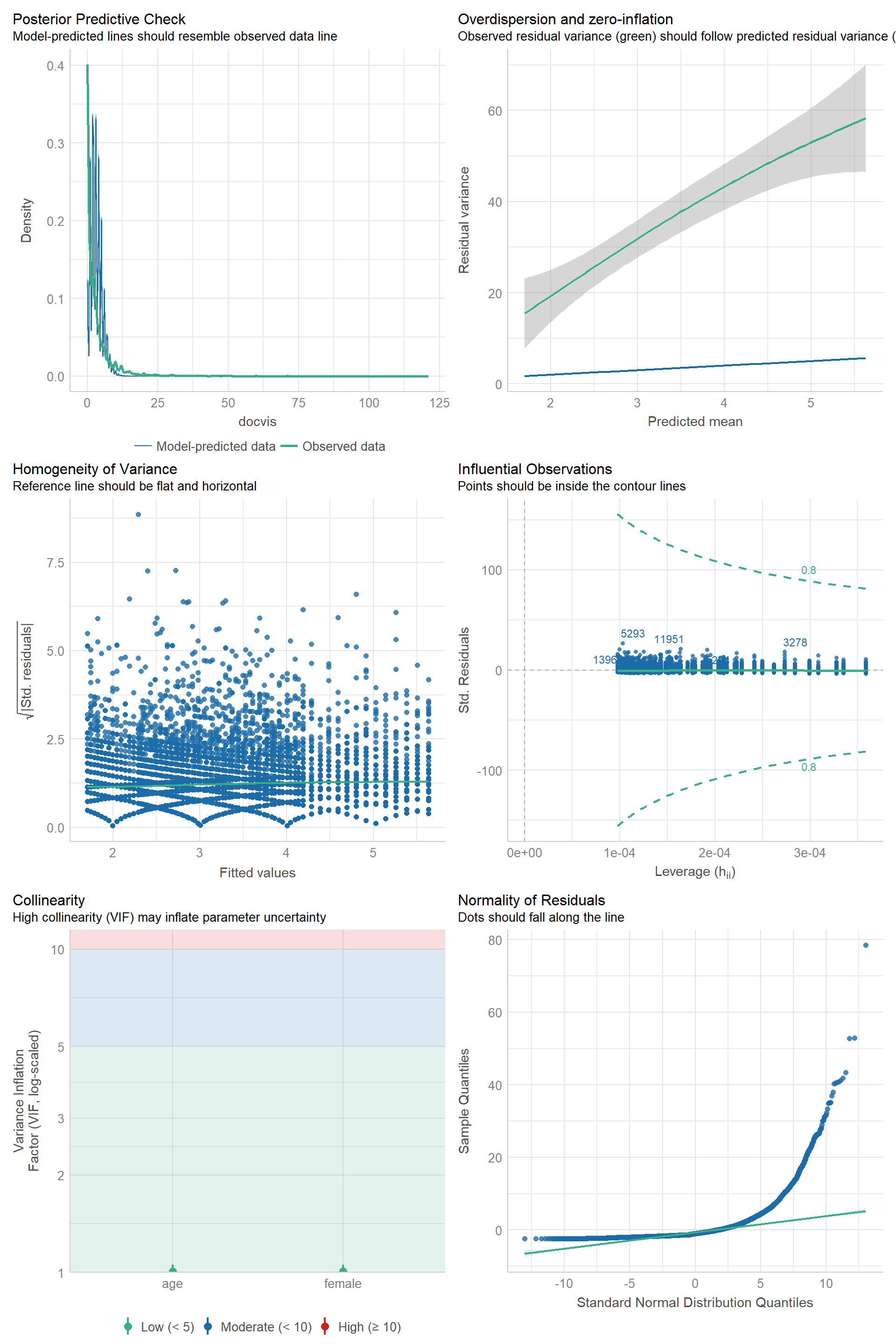
Linealidad del log(yi) respecto a la combinación lineal de predictores.
Observaciones son independientes.
Yi sigue distribución de Poisson.
- Supuesto de equivarianza.
No problemas de regresión:
No puntos influyentes
No colinealidad: Solo cuando esta es un problema.
Supuestos específicos si se busca generalizar a poblaciones conocidas, hacer inferencias causales o ambas.
La regresión Binomial Negativa
Agenda
El Modelo Lineal Generalizado
La regresión de Poisson
La regresión Binomial Negativa
Tablas de regresión reproducibles con {gtsummary}
El modelo de regresión binomial negativa
- Si asumimos que yi tienne un segundo nivel de variabilidad:
yi|λi∼Poisson(λi) y λi∼Gamma(μi,ψ)
Entonces, es posible mostrar que yi sigue una
distribución binomial negativa.Asimismo, el modelo:
yi∼BN(β0+β1x1i+...+βpxip,ψ)
- Componente sistemático:
log(E(yi))=ηi
- Función de enlace:
ηi=β0+β1x1i+...+βpxip
- Componente aleatorio:
yi∼BN(ηi,ψ)
¿Por qué usar binomial negativa y no poisson?
Porque no siempre la variable yi seguirá una distribución de Poisson.
Si sigue una distribución BN, entonces la varianza es mayor a la media (sobredispersion).
La estimación del error estándar deberá tener esto en cuenta.
Caso contrario, sería inválido en dirección anticorservadora.
¿Por qué usar la función de enlace log?
La función de enlace de la BN no es
log(), tampocoidentiy().Sin embargo, se prefiere usar
log()para obtener resultados interpretables:razón de medias.Esto conlleva un problema, no siempre hay convergencia:
Cuando hay sobredispersión, regresión binomial negativa no siempre es la opción factible.
Otra opción puede ser usar regresión quasipoisson o un estimador de varianza robusta.
La regresión BN también retorna razón de medias
La regresión BN permite retornar directamente
razón de medias(RM).Los coeficientes de regresión β del modelo son log(RM), por lo tanto, podemos exponenciarlos para obtener las RM:
β=log(RM)
eβ=RM
glm.nb() paso a paso
- Se usa la función glm.nb() del paquete MASS:
glm.nb() paso a paso
Call:
glm.nb(formula = docvis ~ female + age, data = md_visit, init.theta = 0.4838144807,
link = log)
Deviance Residuals:
Min 1Q Median 3Q Max
-1.5688 -1.3167 -0.4629 0.1127 6.5046
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -0.0366979 0.0457077 -0.803 0.422
female 0.3370406 0.0222361 15.157 <2e-16 ***
age 0.0224236 0.0009913 22.621 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for Negative Binomial(0.4838) family taken to be 1)
Null deviance: 21019 on 19608 degrees of freedom
Residual deviance: 20224 on 19606 degrees of freedom
AIC: 85857
Number of Fisher Scoring iterations: 1
Theta: 0.48381
Std. Err.: 0.00659
2 x log-likelihood: -85849.42400 - Se especifica la ecuación.
glm.nb() paso a paso
| term | estimate | std.error | statistic | p.value |
|---|---|---|---|---|
| (Intercept) | -0.03669795 | 0.0457077355 | -0.8028827 | 4.220425e-01 |
| female | 0.33704062 | 0.0222361245 | 15.1573453 | 6.775303e-52 |
| age | 0.02242362 | 0.0009912903 | 22.6206366 | 2.715341e-113 |
- En el caso de la función de enlace log, los coeficientes son
log(razón de medias), para volverlos interpretables, hay que aplicar antilogaritmo:exp().
glm.nb() paso a paso
| term | estimate | std.error | statistic | p.value |
|---|---|---|---|---|
| (Intercept) | 0.9639673 | 0.0457077355 | -0.8028827 | 4.220425e-01 |
| female | 1.4007960 | 0.0222361245 | 15.1573453 | 6.775303e-52 |
| age | 1.0226769 | 0.0009912903 | 22.6206366 | 2.715341e-113 |
- La función tidy() de {broom} permite hacer esto facilmente:
exponentiate = TRUE.
glm.nb() paso a paso
| term | estimate | std.error | statistic | p.value | conf.low | conf.high |
|---|---|---|---|---|---|---|
| (Intercept) | 0.9639673 | 0.0457077355 | -0.8028827 | 4.220425e-01 | 0.8824872 | 1.053230 |
| female | 1.4007960 | 0.0222361245 | 15.1573453 | 6.775303e-52 | 1.3412238 | 1.463041 |
| age | 1.0226769 | 0.0009912903 | 22.6206366 | 2.715341e-113 | 1.0207472 | 1.024612 |
- También podemos agregar intervalos de confianza.
Casos aplicado
- Identificar factores asociados a que el niño tenga alergia.
- Factores asociados al número de visitas médicas anuales.
- Especificación del modelo
Call:
glm.nb(formula = docvis ~ female + age, data = md_visit, init.theta = 0.4838144807,
link = log)
Deviance Residuals:
Min 1Q Median 3Q Max
-1.5688 -1.3167 -0.4629 0.1127 6.5046
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -0.0366979 0.0457077 -0.803 0.422
female 0.3370406 0.0222361 15.157 <2e-16 ***
age 0.0224236 0.0009913 22.621 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for Negative Binomial(0.4838) family taken to be 1)
Null deviance: 21019 on 19608 degrees of freedom
Residual deviance: 20224 on 19606 degrees of freedom
AIC: 85857
Number of Fisher Scoring iterations: 1
Theta: 0.48381
Std. Err.: 0.00659
2 x log-likelihood: -85849.42400 - Presentación con intervalos de confianza y exponenciada (RM):
# A tibble: 3 × 7
term estimate std.error statistic p.value conf.low conf.high
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 (Intercept) 0.964 0.0457 -0.803 4.22e- 1 0.882 1.05
2 female 1.40 0.0222 15.2 6.78e- 52 1.34 1.46
3 age 1.02 0.000991 22.6 2.72e-113 1.02 1.02female: El número medio de visitas anuales al médico en mujeres fue 20% veces más el de los hombres (RM = 1.40; IC95% 1.34 a 1.46; p < 0.001)age: Por cada incremento de la edad en un año, el número medio de visitas anuales al médico se incrementa en 2.3% (RM = 1.017; IC95% 1.021 a 1.025; p < 0.001).
Linealidad del log(yi) respecto a la combinación lineal de predictores.
Observaciones son independientes.
Yi sigue distribución de Poisson.
- Supuesto de equivarianza.
No problemas de regresión:
No puntos influyentes
No colinealidad: Solo cuando esta es un problema.
Supuestos específicos si se busca generalizar a poblaciones conocidas, hacer inferencias causales o ambas.
Tablas de regresión reproducibles con {gtsummary}
Agenda
El Modelo Lineal Generalizado
La regresión de Poisson
La regresión Binomial Negativa
Tablas de regresión reproducibles con {gtsummary}
Tablas de regresión lineal reproducible
Podemos usar la librería {gtsummary} para esto.
Veamos un ejemplo.
Tablas de regresión lineal reproducible
Podemos reportar la tabla de regreion multivarible de la siguiente manera:
- Primero realizamos el modelo:
# A tibble: 3 × 7
term estimate std.error statistic p.value conf.low conf.high
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 (Intercept) 11.0 0.0505 218. 0 10.9 11.1
2 age 0.0110 0.000341 32.3 5.81e-218 0.0104 0.0117
3 sex -0.474 0.0258 -18.3 8.44e- 74 -0.524 -0.423 Tablas de regresión lineal reproducible
- Se puede crear una tabla de regresión multivariable con la función
tbl_regression()de{gtsummary}:
Tablas de regresión lineal reproducible
- Podemos hacer la tabla de regresiones bivariada con la función
tbl_uvregression()de{gtsummary}:
Tablas de regresión lineal reproducible
- Luego, podemos fusionar ambas tablas en una sola con la función
tbl_merge():
tabla_final <- tbl_merge(list(tabla_univ, tabla_multi), tab_spanner = c("Modelos crudos", "Modelo ajustado"))
tabla_final| Characteristic | Modelos crudos | Modelo ajustado | |||||
|---|---|---|---|---|---|---|---|
| N | Beta | 95% CI1 | p-value | Beta | 95% CI1 | p-value | |
| age | 10,000 | 0.01 | 0.01, 0.01 | <0.001 | 0.01 | 0.01, 0.01 | <0.001 |
| sex | 10,000 | -0.47 | -0.53, -0.42 | <0.001 | -0.47 | -0.52, -0.42 | <0.001 |
| 1 CI = Confidence Interval | |||||||
Tablas de regresión lineal reproducible
- Podemos exportarlo a MS Word para post-procesamiento y reporte:
https://github.com/psotob91
percys1991@gmail.com

R Aplicado a los Proyectos de Investigación - Sesión 9